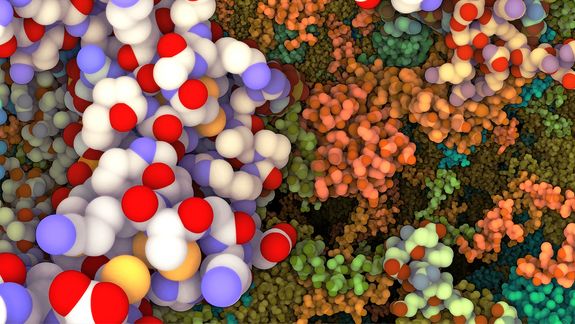
Aus der Ferne sieht ein Virus fast wie ein kleines Kügelchen aus. Kommt man näher, werden komplizierte Strukturen sichtbar, man erkennt einzelne Proteine oder Lipide, bei extremer Vergrößerung gelangt man auf die Ebene einzelner Atome. Gerade die Mikrobiologie liefert ausgezeichnete Beispiele für Objekte, die man auf unterschiedlichen Größenordnungen darstellen muss, um sie verstehen zu können. Diese Aufgabe bringt selbst leistungsfähige Computer an die Grenzen ihrer Möglichkeiten. Am Institut für Computergraphik der TU Wien werden Algorithmen entwickelt, mit denen selbst hochkomplizierte Strukturen, bestehend aus vielen Millionen Einzelteilen, flüssig dargestellt, rotiert und gezoomt werden können. Dafür bekam das TU-Team nun den Austrian Computer Graphics Award (ACGA).
70 Millionen Kugeln
Um die Leistungsfähigkeit der neuen Rechenmethoden unter Beweis zu stellen, hat das Team ein HI-Virus digitalisiert, umgeben von Partikeln, aus denen das menschliche Blut besteht. Atom für Atom sind die Strukturen abgespeichert, der Datensatz besteht aus über 70 Millionen Kugeln. „Die Position all dieser Kugeln am Bildschirm zu berechnen und für jede einzelne Kugel zu bestimmen, ob sie sichtbar ist oder von anderen Kugeln verdeckt wird, wäre völlig unmöglich“, erklärt Prof. Ivan Viola. „Das würde Stunden dauern. Unsere Darstellungsmethode erlaubt es allerdings, am Bildschirm eine Fahrt durch die Strukturen darzustellen – in Echtzeit berechnet, mit 60 Bildern pro Sekunde.“
Möglich wurde das durch eine ganze Reihe neuer technischer Ideen. Kugeln, die zu weit entfernt sind und zu klein erscheinen würden, bleiben von Anfang an unberücksichtigt, manchmal lassen sich mehrere Atome zu einer Kugel zusammenfassen. Der hierarchische Aufbau der Strukturen muss in die Berechnungen einbezogen werden, damit kein wirres Gewimmel winziger Punkte dargestellt wird, sondern übersichtliche, klare Flächen, die erst beim weiteren Heranzoomen ihre Details preisgeben.
Auszeichnung für TU-Projekt
Bei der Computergraphik-Konferenz „PIXEL Vienna“ wurde das Cellview-Tool der TU Wien mit dem Austrian Computer Graphics Award für die „best technical solution“ ausgezeichnet. Zum Computergraphik-Team der TU Wien gehören Ivan Viola, Mathieu Le Muzic, Peter Mindek, Johannes Sorger, Tobias Klein, David Kouril, Brno Nicholas Waldin und Manuela Waldner.
<link https: www.cg.tuwien.ac.at research projects illvisation cellview cellview.php>Hier können Sie selbst einen Virtuellen Spaziergang durch ein Virus wagen
Wer sich für dieses Thema interessiert, kann bei einem Vortrag am 18.11. einen der Pioniere der molekularen Visualisierung kennenlernen: Prof. Arthur Olson wird an die TU Wien kommen. Ort und genauen Zeitpunkt erfahren Sie demnächst auf <link https: www.cg.tuwien.ac.at>www.cg.tuwien.ac.at
Visualisierungen: TU Wien
Daten: Scripps Research Institute, USA vom Molecular Graphics Laboratory, geleitet vom Prof. Arthur Olson